- Acide ribonucléique de transfert
-
Les acides ribonucléiques de transfert, ou ARN de transfert ou ARNt, sont de courts ARN, longs de 70 à 100 nucléotides, qui interviennent lors de la synthèse des protéines dans la cellule. Ce sont des intermédiaires clés dans la traduction du message génétique et dans la lecture du code génétique. Ils apportent les acides aminés au ribosome, la machine cellulaire responsable de l'assemblage des protéines à partir de l'information génétique contenue dans l'ARN messager. Les cellules vivantes contiennent quelques dizaines de sortes d'ARNt, chacune d'elle étant spécifique de l'un des acides aminés.
Sommaire
Rôle dans la traduction du message génétique
Les ARNt sont des acides ribonucléiques simple brin, longs d'environ 70 à 100 nucléotides, que l'on trouve dans le cytoplasme ou dans les organites des cellules vivantes. Ce sont des ARN non codants, transcrits à partir de gènes codés dans le génome. Ils se replient pour adopter une structure complexe comportant plusieurs tiges-boucles.
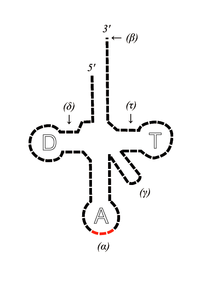
Leur fonction dans la cellule est d'assurer la correspondance entre l'information génétique portée par l'ARN messager et les acides aminés contenus dans la protéine codée par cet ARN messager (ARNm). Ils sont les acteurs clés de la traduction du code génétique. Les ARNt portent l'un des 20 acides aminés attaché par une liaison ester à leur extrémité 3'-OH (β sur la figure 1) et transportent celui-ci au ribosome. Dans une boucle située à l'autre extrémité de la molécule (α sur la figure 1), se trouve une séquence de trois nucléotides, spécifique de l'acide aminé, appelée l'anticodon. L'anticodon s'apparie au codon sur l'ARN messager assurant ainsi la correspondance entre codon et acide-aminé, conformément au code génétique.
Dans la cellule, les ARNt aminoacylés, c'est-à-dire portant un acide aminé estérifié sur leur extrémité 3', s'associent systématiquement en complexe avec le facteur d'élongation EF-Tu (chez les bactéries) ou EF-1 (chez les eucaryotes), lui même complexé au GTP. C'est ce complexe ARNt:EF:GTP, appelé complexe ternaire, qui diffuse dans le site A du ribosome, pour former une interaction codon-anticodon avec l'ARNm. Le ribosome vérifie la complémentarité des bases de l'ARNm et de l'ARNt, puis déclenche l'hydrolyse du GTP lié à EF-Tu. Lorsque celle-ci est réalisée, le ribosome catalyse l'allongement de la chaîne protéique en cours de synthèse (réaction de transpeptidation) et avance sur l'ARN messager. L'ARNt utilisé quitte le ribosome déacylé (avec une extrémité 3'-OH), à l'état libre.
Il est alors rechargé par une enzyme spécifique, appelée aminoacyl-ARNt synthétase, qui catalyse l'estérification de l'acide aminé spécifique. Il existe en général 20 aminoacyl-ARNt synthétases, une pour chaque acide aminé.
Autres rôles des ARNt
En plus de leurs fonctions comme vecteurs des acides aminés dans le mécanisme de traduction des protéines, certains ARNt participent à d'autres processus biologiques. Certains ARNt sont utilisés comme amorces par des transcriptases inverses de rétrovirus ou de rétrotransposons, pour démarrer la synthèse d'un brin d'ADN à partir d'un brin ARN. Le VIH, virus du sida, détourne ainsi l'un des ARNt de la cellule infectée, l'ARNtLys3, pour démarrer son processus de réplication. Le génome viral contient en effet une région complémentaire de cet ARNt cellulaire.
Les ARNt aminoacylés peuvent aussi participer à certaines réactions du métabolisme secondaire, via l'action d'enzymes spécifiques appelées aminoacyl-ARNt transferases, qui catalysent le transfert de l'acide aminé estérifié sur un accepteur protéique ou peptidique. Ces réactions se font indépendamment du ribosome, d'ARNm et sans hydrolyse de GTP. Il peut s'agir de modifications post-transcriptionnelles de protéines, ou bien de synthèse non-ribosomique de peptides bioactifs .
Structure des ARNt
La plupart des ARNt ont une structure canonique, conservée chez toutes les espèces. Ils se replient sur eux mêmes, formant des appariements intramoléculaires de nucléotides pour donner une structure à quatre tiges ou bras, appelée "feuille de trèfle" (figure 1). La tige supérieure, qui porte les extrémités 5' et 3' s'appelle le bras accepteur, car c'est lui qui porte (accepte) l'acide aminé. La tige inférieure, terminée par la boucle de l'anticodon s'appelle bras anticodon, les deux autres tiges s'appellent bras T et bras D (figure 1), car ils portent des ribonucléotides modifiés, autres que A, G, C et U : la ribothymidine (T), pour le bras T et la dihydrouridine (D) pour le bras D.
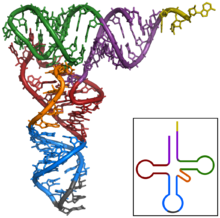
Ces quatre tiges se replient en trois dimensions pour former une structure en forme de "L" (figure 2)[1]. Celle-ci résulte de l'empilement coaxial deux à deux des tiges : le bras T sur bras accepteur et le bras anticodon sur le bras D. Cette structure en forme de "L" est stabilisée par des interactions entre la boucle T et la boucle D, qui fait intervenir des nucléotides modifiés, que l'on retrouve conservés dans la plupart des ARNt. L'extrémité 5' ne présente qu'un seul phosphate. L'extrémité 3' comporte quatre nucléotides non appariés et se termine toujours par le trinucléotide CCA, dont l'adénosine terminale porte la fonction OH où est estérifié l'acide aminé,
Nucléotides modifiés
Les ARNt se caractérisent par la présence d'un grand nombre de nucléotides non-canoniques, ou nucléotides modifiés, dans leur structure. Ces modifications de nucléotides : méthylation, isomérisation, thiolation, réduction... sont incorporées post-transcriptionnellement par des enzymes spécialisées.
On trouve deux grandes classes de nucléotides modifiés dans l'ARNt : ceux qui participent à l'établissement de la stabilisation de la structure tridimensionnelle, par exemple la ribothymidine et la pseudouridine dans la boucle T, et ceux qui sont localisés dans la boucle de l'anticodon et qui interviennent directement dans l'interaction avec l'ARNm et dans la lecture des codons, comme par exemple l'inosine ou ses dérivés.
Certaines modifications de nucléotides sont très simples : ajout d'un groupement méthyle (CH3) et d'autres sont très complexes et nécessitent l'intervention de plusieurs enzymes.
Synthèse
Les ARNt sont synthétisés par transcription à partir de gènes situés dans l'ADN génomique. Chez les bactéries, on trouve fréquemment plusieurs ARNt regroupés sous forme d'opérons[2] qui sont transcrits sous forme d'un seul précurseur qui est ensuite clivé. Chez les eucaryotes, les ARNt sont spécifiquement transcrits par l'ARN polymérase III.
Le transcrit primaire est en général maturé par un certain nombre d'enzymes, qui clivent les extrémités. La ribonucléase P mature l'extrémité 5', et plusieurs ribonucléases sont impliquées dans la maturation du côté 3'. Une enzyme spécifique, l'ARNt nucléotidyl-tranférase ou "CCAse" intervient ensuite pour ajouter ou réparer l'extrémité 3' qui se termine par la séquence CCA.
Les ARNt subissent alors plusieurs étapes de modifications de nucléotides et éventuellement l'épissage d'introns que l'on observe parfois dans certains ARNt eucaryotes..
Codon, anticodon et décodage
Lors du processus de traduction, les trois bases de l'anticodon de l'ARNt s'apparient au codon de l'ARNm. L'interaction entre le premier nucléotide de l'anticodon et le troisième nucléotide du codon est souvent un appariement non-canonique, appelé paire wobble ou "bancale", différente d'un appariement Watson-Crick classique (A-U ou G-C). Cet appariement implique parfois un nucléotide modifié de l'ARNt.
Ces appariements wobble permettent de réduire le nombre d'ARNt nécessaires à la traduction du code génétique, en autorisant la lecture de différent codons synonymes par un seul et même ARNt. Ainsi, l'ARNt spécifique de l'isoleucine dans la souris à un anticodon IAU, où I est une inosine qui peut s'apparier aux trois codons AUU, AUC et AUA, en formant des paires I-U, I-C et I-A.
ARNt et aminoacyl-ARNt synthétase
L'estérification de l'acide aminé spécifique à l'extrémité 3'-OH de l'ARNt est une étape clef du décodage du code génétique. On appelle cette étape l'aminoacylation et, dans toutes les cellules vivantes, il existe une vingtaine d'enzymes, les aminoacyl-ARNt synthétases, dont la fonction est de catalyser cette étape.
Chacune de ces enzymes est spécifique d'un acide aminé donné et reconnait le ou les ARNt correspondant. Ces enzymes sont capables de discriminer les ARNt spécifiques de son acide aminé, en reconnaissant des motifs de nucléotides particuliers, souvent dans l'anticodon lui-même, mais parfois dans d'autres régions de l'ARNt. Cette reconnaissance est cruciale, car il n'y a plus de contrôle qualité ultérieur au niveau du ribosome : toute erreur dans l'aminoacylation se traduira donc ensuite par une erreur dans le décodage du message génétique.
L'aminoacylation comprend deux étapes: l'activation de l'acide aminé par adénynylation et la formation du lien ester entre l'acide aminé et l'hydroxyle 2' ou 3' du ribose du nucléotide 3' de l'ARN de transfert.
La première étape consiste en l'attaque nucléophile de l'acide aminé sur une molécule d'ATP reconnue par l'aminoacyl-ARNt synthétase. L'oxygène de cet acide aminé brisera le lien entre le premier et le second des trois phosphates de l'ATP. Ainsi, il se formera un nouveau lien, un lien anhydride, entre l'acide aminé et l'AMP (adénosine monophosphate) ainsi généré. L'acide aminé est ainsi activé sous forme d'aminoacyl-adénylate, qui reste lié à l'enzyme.
Deuxième étape: catalyse d'un lien ester par l'ARNt. En utilisant l'énergie contenue dans l'aminoacyl-adénylate, l'ARN de transfert fera une attaque nucléophile sur l'acide aminé. Cela créera un lien ester, qui sera encore riche en énergie.
Après ces deux étapes, l'ARN de transfert est prêt à amener son acide aminé spécifique vers le ribosome et l'ARN messager[3].
Historique
L'existence des ARNt, comme « adapteurs » entre les acides aminés et l'ARN messager, a été postulée par Francis Crick[4], avant que ceux-ci ne soient effectivement découverts par Hoagland et Zamecnick en 1958[5]
C'est également Francis Crick qui a formulé l'hypothèse de « l'interaction bancale » ((en) wobble hypothesis) qui permet d'expliquer le fait qu'un ARNt donné puisse lire plusieurs codons synonymes[6].
Notes et références
- (en) J.D. Robertus, J.E. Ladner, J.T. Finch, D. Rhodes, R.S. Brown, B.F. Clark et A. Klug, « Structure of yeast phenylalanine tRNA at 3 A resolution. », dans Nature, vol. 250, 1974, p. 546–551 [lien PMID], (en) S.H. Kim, F.L. Suddath, G.J. Quigley, A. McPherson, J.L. Sussman, A.H. Wang, N.C. Seeman et A. Rich, « Three-dimensional tertiary structure of yeast phenylalanine transfer RNA. », dans Science, vol. 250, 1974, p. 546–551 [lien PMID]
- (en) Maurille J. Fournier et Haruo Oseki, « Structure and organization of the transfer ribonucleic acid genes of Escherichia coli K-12 », dans Microbiol. Rev., vol. 49, 1985, p. 379-287 [lien PMID]
- H. Lodish, A. Berk, P. Matsudaira, C.A. Kaiser, M. Krieger, M.P. Scott, L. Zipursky et J. Darnell, Biologie moléculaire de la cellule, Bruxelles, de Boeck, 2005, 3e éd. (ISBN 978-2804148027)
- (en) Francis H. Crick, « On protein synthesis », dans Symp. Soc. Exp. Biol., vol. 12, 58, p. 138-163 [texte intégral, lien PMID]
- (en) M.B. Hoagland, M.L Stephenson, J.F. Scott, H.I. Hecht et P.C. Zamecnik, « A soluble ribonucleic acid intermediate in protein synthesis », dans J. Biol. Chem., vol. 231, 1958, p. 241-257 [lien PMID]
- (en) F.H. Crick, « Codon--anticodon pairing: the wobble hypothesis. », dans J. Mol. Biol, vol. 19, 1966, p. 548-555 [lien PMID]
Voir aussi
Articles connexes
- ARN
- ARN messager
- ADN
- Une liste des différents types d'ARN
- Liste d'abréviations de biologie cellulaire et moléculaire
Liens externes
- (en) Compilation des séquences d'ARNt à l'Université de Bayreuth
- (en) G~tRNA~db: The Genomic tRNA Database
- Portail de la biologie cellulaire et moléculaire
Wikimedia Foundation. 2010.